Di Vittoria Brambilla per SIGA – Società Italiana Genetica Agraria
Le tecniche di genome editing vengono utilizzate in diversi ambiti della scienza e hanno enormi potenzialità per l’agricoltura. Infatti, il genome editing permette di inserire piccoli cambiamenti all’interno dei genomi che possono ricreare alleli favorevoli nelle piante di interesse agrario.
CRISPR, la tecnica biomolecolare più versatile
Il genome editing è basato su tecniche biomolecolari.
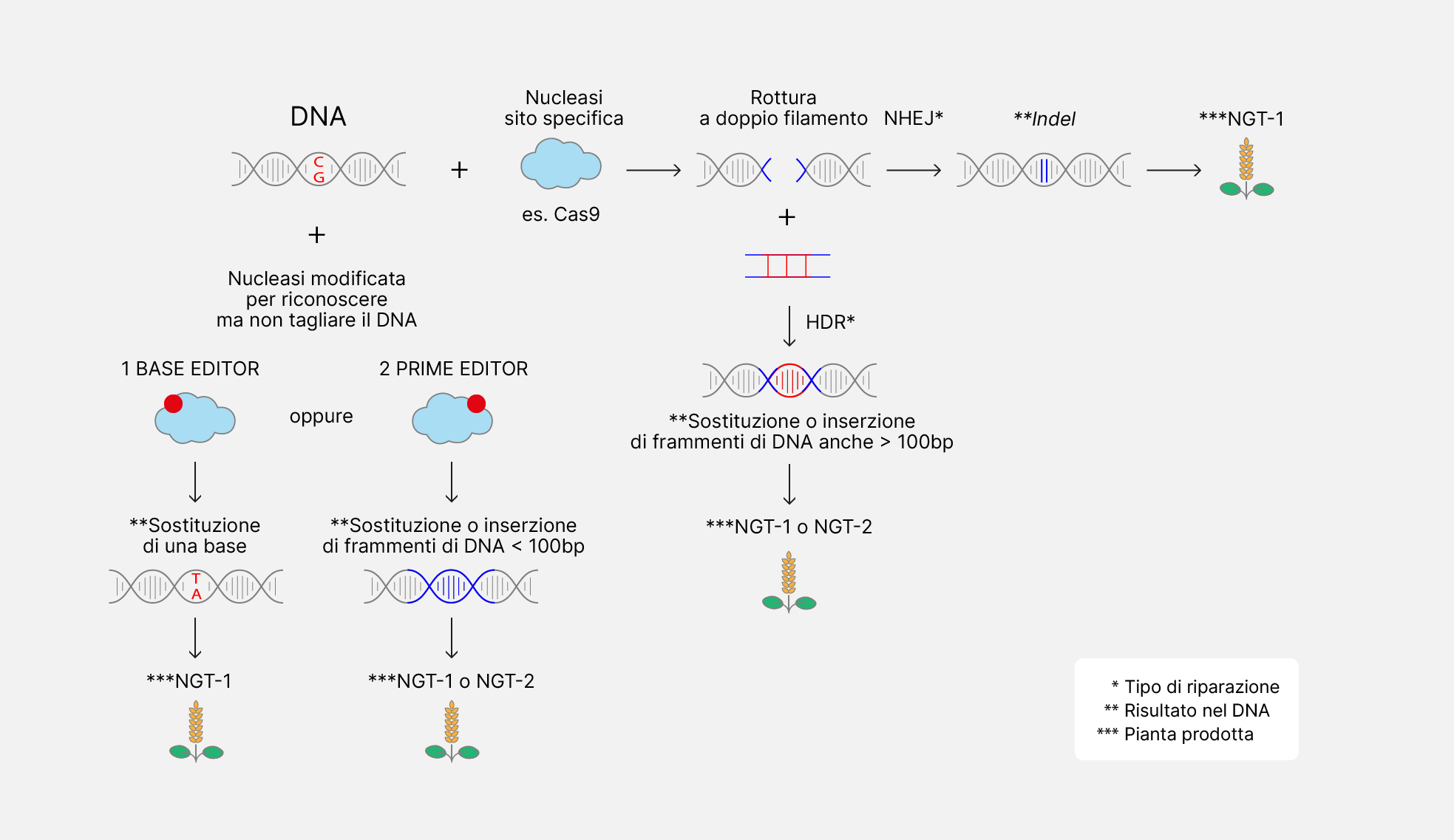
La più nota e versatile, ad oggi, è la tecnica CRISPR, messa a punto nel 2012. Essa permette di tagliare il doppio filamento del DNA e di innescare due possibili vie di riparazione cellulare: la Non Homologous End Joining (NHEJ) e la Homology-Directed Repair (HDR).
L’enzima che taglia il DNA è una nucleasi sito-specifica e le due tipologie di riparazione portano a risultati differenti: la NHEJ inserisce frequentemente mutazioni indel (con questo termine ci riferiamo a inserzioni o delezioni molto piccole, di 1 o poche basi) che sono solitamente inserite per ottenere la perdita di funzione dei geni, mentre la HDR può essere provocata causando due tagli sul DNA a breve distanza e fornendo del DNA stampo che può essere usato per la riparazione della rottura grazie a sequenze di omologia esterne alla regione spaccata.
La differenza sostanzialmente è che, sfruttando la NHEJ ci si limita all’inserzione di indel, mentre tramite DNA stampo e HDR si può virtualmente inserire qualsiasi sequenza di DNA in modo sito specifico. Pertanto, le modifiche risultanti sono di entità diversa.
Base editing e prime editing
Quando le mutazioni da inserire devono essere specifiche, oltre alla HDR si può scegliere di ricorrere ad altre due evoluzioni della tecnica CRISPR, dove la nucleasi è stata modificata per riconoscere il DNA ma non tagliarlo.
La prima è il base editing, che è in grado di sostituire in modo specifico singole basi di DNA (ad esempio una Citosina in una Timina o una Adenosina in una Guanina).
L’altra è il prime editing, che permette di sostituire o solo inserire sequenze di DNA a piacimento. Dunque, se il base editing permette di inserire specifici polimorfismi a livello delle singole basi (SNPs), anche se in modo più specifico del NHEJ, il prime editing può essere usato per sostituire o aggiungere sequenze più lunghe.
Specie agrarie: tante possibilità di miglioramento genetico
Nelle specie agrarie, soprattutto in quelle ben caratterizzate come i cereali e alcune orticole, per molti geni sono state mappate negli anni varianti alleliche favorevoli per tratti legati al miglioramento genetico. Queste sono talvolta originate da indels, che causano la perdita di funzione di un gene, da SNPs specifici oppure da cambiamenti più grandi. Dunque, per poterle riprodurre tramite genome editing sarà necessario utilizzare approcci diversi: a volte basterà lasciare che la cellula ripari il suo DNA tramite NHEJ, altre volte sarà necessario ricorrere a tecniche più sofisticate come l’attivazione della HDR, il base editing o il prime editing. Se la prima tecnica è molto semplice e di facile ottenimento, le altre hanno una efficienza più bassa e maggiori restrizioni al loro utilizzo.
Il genome editing per ricerca di base o applicata per rendere il riso più resistente a una malattia
In laboratorio, nella maggior parte dei casi usiamo genome editing e NHEJ per inattivare i geni ma recentemente abbiamo anche inserito varianti tramite HDR, base editing e prime editing.
NHEJ è quello che ci serve tutte quelle volte che vogliamo capire la funzione di un gene perché, eliminandolo, possiamo vedere cosa accadrebbe alla pianta in sua assenza, ma è anche utile quando il fenotipo è dato dalla mancanza della funzione di uno o più geni. Nella letteratura scientifica si trovano molti geni interessanti che migliorano il fenotipo della pianta se inattivati. Uno di questi è il gene di suscettibilità alla malattia fungina del riso nota come “brusone”, che si chiama Pi21. Delezioni e anche mutazioni inattivanti in Pi21 causano una maggior resistenza a brusone.
Nel genoma di riso ci sono altri geni che hanno probabilmente una funzione simile a Pi21. Due di questi sono HMA1 e HMA2 che, se inattivati, migliorano la resistenza a brusone. La nostra prima applicazione di genome editing e NHEJ a scopo di miglioramento genetico risale a sei anni fa e ci ha portato ad ottenere delle piante di riso meno suscettibili a brusone tramite inattivazione di Pi21, HMA1 e HMA2.
Ora serve la sperimentazione in campo
Le applicazioni del genome editing a scopo di ricerca di base non suscitano grandi problemi visto che le piante ottenute solitamente servono a fare esperimenti in laboratorio. Ma è fondamentale che le piante prodotte per il miglioramento genetico siano sperimentate in campo, dove poi potranno essere coltivate se si comporteranno in modo migliore delle loro corrispettive non genome edited. Per questo motivo, una volta ottenute piante dove Pi21, HMA1 e HMA2 sono stati inattivati tramite l’introduzione di indels indotte da NHEJ, abbiamo cercato di sperimentarle in campo. Nonostante HDR e prime editing possano essere utilizzati per introdurre nel DNA delle piante sequenze di DNA che vengono da altri organismi o sono addirittura sintetiche, la maggior parte delle piante genome edited, tra cui le nostre resistenti a “brusone”, contengono indels del tutto simili a quelle che si trovano comunemente in natura.
Ma la scienza corre molto più veloce delle normative.
Visto che le applicazioni della biologia molecolare in agricoltura sono tra quelle viste con maggior sospetto dai non esperti, una sentenza della Corte di Giustizia Europea del luglio 2018 ha preso una posizione cautelativa concludendo che il genome editing avrebbe dovuto essere normato alla stregua degli OGM. Una sentenza di morte per il genome editing, visto che gli OGM non possono essere coltivati sulla maggior parte del suolo comunitario e non possono essere nemmeno seminati per scopi sperimentali in Italia.
Il punto sull’attualità
Col passare del tempo, complice la deregolamentazione progressiva da parte degli stati extraeuropei rispetto alle piante prodotte con genome editing, le pressioni degli scienziati e degli agricoltori che chiedevano nuove soluzioni hanno spinto la Commissione Europea a consultare esperti, portatori di interesse e cittadini per proporre, infine, a luglio 2023, una possibile normativa che separi OGM da genome editing.
Le piante fatte con NHEJ o base editing e quelle in cui HDR o prime editing non hanno permesso di introdurre sequenze più lunghe di 20 nucleotidi vengono inserite nella categoria NGT-1 (New Genomic Techniques tipo 1) mentre il tipo 2 rimane escluso dalla proposta. La proposta della Commissione è stata approvata anche dal Parlamento Europeo nel febbraio 2024. Ora manca un passaggio in Consiglio con l’opinione dei singoli stati membri ma siamo sempre più vicini ad una nuova normativa che sia basata sul tipo di prodotto rispetto al processo con cui è stata fatta.
Nel frattempo, alcuni stati hanno cercato di favorire la sperimentazione in campo di piante NGT-1 tra cui l’Italia che ha citato le sperimentazioni in campo di queste piante, chiamate in Italia anche TEA (o frutto delle “Tecniche di Evoluzione Assistita”) nella legge per contrastare la siccità del giugno 2023 e che ha snellito le pratiche per poter richiedere campi sperimentali.
Grazie a questa legge, che però ha validità solo fino a fine 2024, abbiamo fatto richiesta al Ministero dell’Ambiente e della Sicurezza Energetica italiano di poter fare un campo sperimentale del nostro riso NGT-1 con indels in Pi21, HMA1 e HMA2 in primavera-estate 2024. La nostra richiesta è arrivata esattamente 20 anni dopo l’ultima richiesta, bocciata, per mettere in campo limoni OGM dall’Università di Catania. Mentre aspettiamo una decisione del Ministero altri gruppi di ricerca italiani stanno seguendo la nostra strada.